Lebenswissenschaften – Gruppe von Thomas Sokolowski
Forschungsschwerpunkt
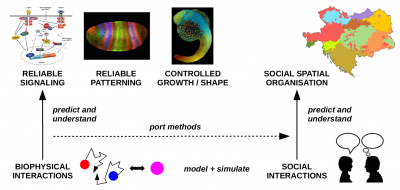
Unsere Kernfragen lauten: Wie können Zellen zuverlässig biochemische Signale generieren, empfangen, verarbeiten und weitergeben trotz beschränkter molekularer Ressourcen und damit einhergehendem starken biologischen Rauschen? Wie können Gewebe und ganze Organismen sich nahezu fehlerfrei entwickeln und reproduzieren, wenn ihre biologische Entwicklung auf solcher Art verrauschten Prozessen basiert? Welche biophysikalischen Mechanismen hat die Evolution hervorgebracht, um biologischen Zellen zu ermöglichen, biologisches Rauschen zwecks zuverlässiger Informationsverarbeitung zu kontrollieren? Welchen Einfluss haben darauf insbesondere räumliche Effkte, wie Moleküldiffusion und Transport von Molekülen auf dem Zytoskelett? Und schließlich: Können wir unser mechanistisches Verständnis von Mechanismen, die biologisches Rauschen in Organismen kontrollieren, auch dazu verwenden, stochastische Prozesse auf der Skala von vielen interagierenden Organismen und Populationen besser zu verstehen?
Diese Fragestellungen erforschen wir mit Hilfe einer Kombination von Methoden aus der statistischen und Computer-Physik, mit einem besonderen Fokus auf biophysikalisch realistische und gleichzeitig recheneffiziente räumlich-stochastische Simulationen. Das Kernstück unseres Ansatzes bildet daher die Entwicklung ereignisgetriebener räumlich-stochastischer Algorithmen, welche ihre hohe Recheneffizienz der Verwendung mathematischer Zufallsverteilungen für zukünftige Simulationsereignisse verdanken. Um hierfür zuverlässige Voraussagen machen zu können, müssen die verwendeten Verteilungen sorgfältig aus den zu Grunde liegenden biophysikalischen und biochemischen Grundprinzipien hergeleitet werden. Zusätzlich zu diesem Ansatz verwenden wir numerische Optimierungsalgorithmen, um in den (üblicher Weise stark ausgedehnten) Parameterräumen unserer Modelle diejenigen Parameter-Regimes zu finden, welche eine effektive Kontrolle des biologischen Rauschens garantieren können. Unsere Herangehensweise kombiniert daher theoretische Herleitungen mit modernen Techniken des Hochleistungs-Rechnens.
Interessiert?
Studierende, die daran interessiert sind, physik-basierte rechnerische Ansätze zur Ergründung der Robustheit biologischer Systeme anzuwenden, sind herzlich eingeladen, uns zu kontaktieren um gemeinsam Möglichkeiten für Projekte oder Praktika auszuloten!
Studierende, die an der Teilnahme an den von uns an der Goethe-Universität angebotenen Kursen und Seminaren in den Bereichen Biophysik, Computerphysik und Wissenschaftliche Programmierung interessiert sind, finden nähere Informationen dazu auf der Seite "Lehre".
Aktuelle Forschungsprojekte
GEWEBEENTWICKLUNG IN FLIEGEN- UND MAUSEMBRYOS
Einer der faszinierendsten biologischen Prozesse ist die Entwicklung eines Embryos gemäß seines genetischen "Bauplans" aus einer anfangs einzigen befruchteten Zelle. Obwohl die grundlegenden Schritte der Embryoentwicklung heute fast schon zum Allgemeinwissen gehören, wird oft übersehen, dass die Embryoentwicklung auf der mikroskopischen Ebene auf biochemischen Prozessen basiert, welche mit einer oft stark begrenzten Anzahl an Molekülen auskommen müssen, und daher von Grund auf stochastisch sind. Dies führt zu starkem biologischen Rauschen in den proteinbasierten Signalen, welche die frühe Zelldifferenzierung in sich entwickelndem Gewebe steuern, und kontrastiert dabei mit der beobachteten außerordentlich hohen Präzision und Reproduzierbarkeit von Embryoentwicklungsprozessen. Darüber hinaus wird diese hohe Präzision in vielen Systemen in so kurzer Zeit erreicht, dass eine einfache zeitliche Mittelung der verrauschten Proteinsignale als simple Maßnahme zur Unterdrückung des Rauschens bei weitem nicht ausreicht. Wie also kann frühe Embryoentwicklung derartig zuverlässig und schnell vonstattengehen trotz beschränkter molekularer Ressourcen?
Unsere Forschung zielt darauf ab, biophysikalische und biochemische Mechanismen zu ergründen, welche das biologische Rauschen in Gewebeentwicklungsprozessen unter Kontrolle halten und somit eine zuverlässige, reproduzierbare Entwicklung garantieren. Dazu entwickeln wir biophysikalisch realistische räumlich-stochastische Modelle sich entwickelnder Embryos in ihren frühen Stadien, und bringen die rechnerische Effizienz dieser Modelle auf ein hohes Niveau mithilfe von Coarse-Graining-Techniken und effizienten (zuallermeist ereignisgesteuerten) Simulationsalgorithmen. Diese Herangehensweise erlaubt es uns, in den oft durch naive Methoden nicht erschließbaren großen Parameterräumen der zu Grunde liegenden räumlich-stochastischen Modelle diejenigen Parameter-Regimes zu identifizieren, welche das biologische Rauschen in Entwicklungsprozessen optimal unter Kontrolle halten.
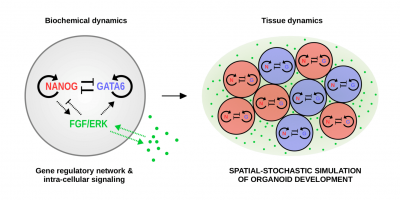
Momentan liegt unser Fokus auf zwei spezifischen Systemen: auf dem "Gap Gene System" in der frühen Embryoentwicklung der Fruchtfliege Drosophila Melongaster, und auf der frühesten Entwicklung des Mausembryos vor seiner Einnistung. Obwohl diese zwei Systeme auf Genregulations-Motiven aufbauen, welche in Embryosystemen immer wieder auftauchen, unterscheiden sie sich grundlegend in der Abfolge der entwicklungsbiologischen Schritte: Während im frühen Fliegenembryo zunächst eine homogene, aus mehreren Tausend Zellen bestehende Gewebestruktur entsteht, deren unterschiedliche Zellentypen erst in einem nachfolgenden Schritt durch das auslesen räumlicher Proteingradienten spezifiziert werden, erfolgt die früheste Entwicklung des Mausembryos äußerst dynamisch, so dass unterschiedliche Zelltypen hier bereits während der Zellteilung und -reorganisation spezifiziert werden. Durch den Vergleich der unterschiedlichen Mechanismen zur Kontrolle des biologischen Rauschens in beiden Systemen zielen wir darauf ab, generische, in vielen Systemen wiederkehrende Strategien zur zuverlässigen Zell-Spezifizierung von denjenigen zu unterscheiden, welche unter den jeweils speziellen Bedingungen in spezifischen Organismen auftreten, und somit zu verstehen, welche dieser Mechanismen unter welcher Art von Randbedingungen vorteilhaft sind.
RÄUMLICHE ASPEKTE IN BINDUNGSPROZESSEN VON TRANSKRIPTIONSFAKTOREN
Transkriptionsfaktoren sind Proteine, die durch Anbinden an designierte DNA-Regionen die Expression von Genen gezielt aktivieren oder supprimieren können, und formen somit die Basis aller Genregulationsprozesse. In den vergangenen Jahrzehnten haben die Lebenswissenschaften sehr viel gelernt über die Prinzipien der Genregulation durch Transkriptionsfaktoren, über die grundlegend stochastische Natur dieser Prozesse und über zahlreiche Mechanismen, welche diese "in Schach halten". Dennoch bleibt unser Wissen darüber, wie die Regulierung von Genexpression trotz begrenzter molekularer Ressourcen zuverlässig ablaufen kann, unvollständig. Dies gilt insbesondere für eukaryiontische Systeme, in welchen die durch Transkriptionsfaktoren regulierten DNA-Bereichen häufig eine komplexere Struktur aufweisen als es notwendig schiene, z.B. durch das Vorhandensein mehrerer identischer Bindungsregionen oder von vergleichsweise weit von der eigentlichen Promoter-Region entfernten "Enhancer-Regionen". Zudem scheinen neuere Forschungsergbnisse auf eine bisher wenig beachtete Art von Regulationsmechanismus hinzuweisen, bei der, im Gegensatz zum klassischen "search and bind" Mechanismus, dynamische Ensembles von Transkriptionsfaktoren ("transcription factor hubs") in der nähe der relevanten DNA-Zielregionen akkumulieren und daran kurz aber wiederholt anbinden. Es ist nicht endgültig geklärt, inwiefern ein solcher Mechanismus dem klassischen ("search and bind") Mechanismen, in welchen einem längeren Suchprozess eine starke Anbindung erfolgt, überlegen sein könnte.
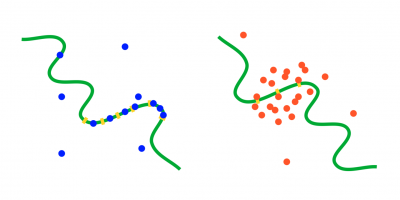
Wir erforschen wie bestimmte Verteilungen von Transkriptionsfaktor-Bindungsstellen auf der DNA die Anbindungswahrscheinlichkeit beeinflussen, und auch wie dynamische "transcription factor hubs" überhaupt einen wohldefinierten Regulationszustand auf der DNA definieren können. Wir ergründen auch, worin die Vor- und Nachteile solcher "komplexerer" Bindungsmechanismen bestehen im Vergleich zu den klassischen "search and bind" Prozessen. Dazu kombinieren wir teilchenbasierte stochastische Simulationen (v.a. eGFRD2, einen ereignisgesteuerten teilchenbasierten Simulator für Reaktions-Diffusions-Prozesse) mit mathematischer Modellierung.
RÄUMLICH-STOCHASTISCHE PROZESSE IN ORGANISMENPOPULATIONEN
In den vorangehenden Jahrzehnten haben sowohl die biochemischen Simulationstechniken als auch unser Verständnis von räumlich-stochastischen Effekten in biologischen Zellen und Geweben große Fortschritte gemacht. Allerdings haben räumlich-stochastische Effekte, und hier insbesondere die Diffusion, eine ähnlich große Bedeutung für stochastische Prozesse auf der Skala von Populationen. Wichtige Beispiele hierfür sind: die Ausbreitung von Sprachen oder anderen sozialen "Merkmalen", und Epidemien. Dadurch, dass in diesen Prozessen die Erzeugung, das Verschwinden / die Degradierung und der Transport von relevanten "Informationsträgern" auf stochastischen Prozessen beruht, haben sie eine starke Ähnlichkeit mit räumlich-stochastischen biochemischen Prozessen. Ausgehend von unserer Expertise im Bereich der biochemischen Modellierung und Simulation ergründen wir daher, inwiefern unsere Methoden und Erkenntnisse aus dem Bereich der Zellbiologie sich auf vergleichbare Systeme und Prozesse auf der Populationsskala übertragen lassen, um unser Verständnis der dort zu Grunde liegenden stochastischen Prozesse und zugehöriger Kontrollmechanismen zu erweitern.